2021. 6. 26. 12:03ㆍR 공부/비지도학습
PCA는 내가 가진 데이터에서 가장 중요한 성분을 순서대로 추출하는 기법이다.
- 내 데이터의 분산을 가장 잘 설명해주는 축이 주성분1(PC1)
- PC1에 직교하는 축이 주성분2(PC2)

이미지 데이터에서 사용하는 PCA





공분산 행렬은 데이터 간 퍼져있는 정도를 나타내는 행렬
PCA는 분산을 최대화 하는축(주성분)을 찾는 작업이다
공분산행렬

Eigenvalue(고유값) & Eigenvector(고유벡터)
공분산 행렬에서 나타나는 고유한 벡터와 벡터의 고유값을 의미한다.
고유한 벡터: 분산의 방향, 주성분
벡터의 고유값: 분산의 크기, 주성분의 연산


v는 0이 아니어야한다. I는 단위행열이다. 역행렬이 존재하면 안되는 걸 찾는것.
Eigenvalue의 크기 순서대로 eigenvector을 나열한다.
정렬된 eigenvector 중 필요한 만큼 일부 선택하여 차원축소를 한다.
필요한 만큼이 얼마일까?
주성분의 개수설정하는것을 알아보자 3가지 방법이있다.
- 시각화를 위해 2 or 3 개로 주성분 개수를 설정한다.(2차원 or 3차원) = 3차원 이상이면 시각화할수가 없다.
- Eigenvalue>1을 기준으로 주성분 개수를 설정한다.
- 주성분 개수가 늘어나도 분산이 더이상 추가되지 않는 지점에서 주성분 개수를 설정한다.
이제 실습을 해보자
오늘 사용할 데이터는 머신러닝에서 예제로 자주 사용되는 iris 꽃 데이터이다.
데이터를 살펴보자
head(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
colSums(is.na(iris))colSums 함수 = 각 변수별로
is.na함수 = 결측치가 있는지
summary(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100 setosa :50
1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300 versicolor:50
Median :5.800 Median :3.000 Median :4.350 Median :1.300 virginica :50
Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500 species는 범주형 변수라서 카운트 된 값이 나온다.
보통 데이터를 볼때 Median과 Mean를 주로 보는데 둘의 차이가 많으면 outlier 값이 많은것이기 때문!
boxplot(iris[,1:4])데이터의 형태를 알아보기 위해 boxplot을 이용해서 나타낸다.

species는 범주형이기 때문에 제외하고 나타냈다.
iris.pca <- prcomp(iris[1:4],center = T, scale. = T) #PCA 함수pca 함수는 한줄이면 된다.
iris데이터의 1열부터 4열 까지, center = T, scale = T 은 평균은 0 분산은 1로 표준화를 해줘라 라는뜻이다.
summary(iris.pca)
Importance of components:
PC1 PC2 PC3 PC4
Standard deviation 1.7084 0.9560 0.38309 0.14393 #표준편차 표준편차를 제곱하면 분산 = eigenvalue
Proportion of Variance 0.7296 0.2285 0.03669 0.00518 #분산이 전체분산에서 차지하는 비율
Cumulative Proportion 0.7296 0.9581 0.99482 1.00000 #누적분산의 비율
iris.pca$rotation #각 주성분의 eigenvector
PC1 PC2 PC3 PC4
Sepal.Length 0.5210659 -0.37741762 0.7195664 0.2612863
Sepal.Width -0.2693474 -0.92329566 -0.2443818 -0.1235096
Petal.Length 0.5804131 -0.02449161 -0.1421264 -0.8014492
Petal.Width 0.5648565 -0.06694199 -0.6342727 0.5235971
eigenvector = 각 변수들의 가중치
head(iris.pca$x,10) #각 주성분의 값
PC1 PC2 PC3 PC4
[1,] -2.257141 -0.47842383 0.12727962 0.024087508
[2,] -2.074013 0.67188269 0.23382552 0.102662845
[3,] -2.356335 0.34076642 -0.04405390 0.028282305
[4,] -2.291707 0.59539986 -0.09098530 -0.065735340
[5,] -2.381863 -0.64467566 -0.01568565 -0.035802870
[6,] -2.068701 -1.48420530 -0.02687825 0.006586116
[7,] -2.435868 -0.04748512 -0.33435030 -0.036652767
[8,] -2.225392 -0.22240300 0.08839935 -0.024529919
[9,] -2.326845 1.11160370 -0.14459247 -0.026769540
[10,] -2.177035 0.46744757 0.25291827 -0.039766068plot(iris.pca, type = 'l', main = 'Scree Plot') #PC의 분산을 y축으로 scree plot 생성주성분의 개수를 선택하기 위해 Scree Plot을 그려본다.
옵션중에 type = 'l' 은 line을 의미한다.

여기서 elbow point는 3이다.
하지만 variances = eigenvalue 가 1보다 커야하므로 2일때가 적당하다.
head(iris.pca$x[,1:2], 10) #2열까지 가져오며 2차원으로 축소
시각화를 해보자
library(ggfortify)
autoplot(iris.pca,data = iris, colour = 'Species')
autoplot에 data = iris, colour 을 넣은건 레이블을 같이 시각화 해줘서 확인하기 위함이다.

빨간색 종은 다른 종들과 뚜렷하게 구분이되는 종이다.
이번엔 사진 분석을 PCA를 통해 실습해보자

오늘 사용할 사진은 귀여운 고양이 사진이다.
library(jpeg)
cat <- readJPEG('cat.jpg')
class(cat)
class를 확인해보면 array라고 나온다.
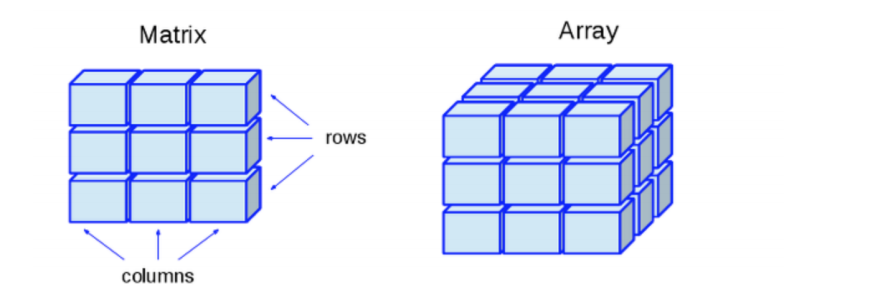
array는 matrix를 몇겹 겹쳐놓은 3차원 데이터이다.
R/G/B가 들어있으므로 3차원이다.
dim(cat)
[1] 360 480 3 # R G BR G B 를 각자 하나씩 빼면 2차원으로 된다.
r <- cat[,,1] #R에 해당하는데이터
g <- cat[,,2] #G에 해당하는데이터
b <- cat[,,3] #B에 해당하는데이터
cat.r.pca <- prcomp(r,center = F) #r 데이터 주성성분 분석
cat.g.pca <- prcomp(g,center = F) #g 데이터 주성성분 분석
cat.b.pca <- prcomp(b,center = F) #b 데이터 주성성분 분석
rgb.pca <- list(cat.r.pca,cat.g.pca,cat.b.pca) #분석결과 rgb로 합침
pc <- c(2,10,50,100,300) #축소할 차원수
for (i in pc) {
pca.img <- sapply(rgb.pca, function(j){ #sapply는 리스트의 각각데이터에 함수를 적용해줌, funtion(j)에서 j = rgb.pca가된다.
compresssed.img <- j$x[,1:i] %*% t(j$rotation[,1:i]) #주성분의 값 x , rotation = eigenvector 이고 %*%은 행렬곱이다.
},simplify = 'array' #array로 만들어줘라
)
writeJPEG(pca.img,paste('cat_pca_',i,'.jpeg',sep = ' ')) #jpeg 파일로 저장한다.
}





300차원이 되고나서야 우리가 처음넣은 사진이랑 비슷하게 나온다.
'R 공부 > 비지도학습' 카테고리의 다른 글
| Hieararchical Clustering Analysis (0) | 2021.06.28 |
|---|---|
| k-Means Analysis (0) | 2021.06.27 |